お知らせ
- 2018.11.05
jMorpのゲノム情報を拡張~INDEL頻度情報などを新たに公開~
東北メディカル・メガバンク機構(ToMMo)では、公開してきたゲノム解析情報の更なる拡充を行い、約3,500人分の全ゲノム解読情報に対して、①INDEL頻度情報、②1塩基ごとの平均深度データ情報、③変異情報をタンパク質立体構造上にマッピングするツール、などを追加すると共に、当計画で行ったすべての全ゲノム解読(約4,000人分)に対して解析データの有無を整理した④ゲノム解析レポジトリ情報、を新たに公開しました。本公開は、ToMMoがゲノム解析情報やメタボローム解析情報を公開するプラットフォームとしてきた、日本人多層オミックス参照パネル(jMorp: Japanese Multi Omics Reference Panel)で行われました。より多層的で情報量の多いデータベースが構築されたことにより、多くの研究者により利便性高く利活用されることが期待されます。
ToMMoは、コホート調査に参加された方々からのDNA試料をもとに全ゲノム解析を行い、日本人の全ゲノムリファレンスパネルの構築に取り組んできました。この全ゲノムリファレンスパネルは、2015年の約1,000人の解析を行ったバージョン(1KJPN)から、2016年には約2,000人(2KJPN)、2017年には約3,500人(3.5KJPN)へと順次サイズを拡大しています。その間、当機構においては、データ解析手法の進歩に対応し、国際標準に準拠した手法に更新、海外の大規模ゲノム解析との比較がより容易なリファレンスパネルとして再構築を進め、2018年6月には、更にミトコンドリアやX染色体の解析を行った結果を追加するなど内容を大幅に拡充・更新した3.5KJPNv2を公開しました。3.5KJPNv2以降のゲノム情報はjMorpにて公開しています。
今回、jMorpをさらに拡張し、INDEL頻度情報をはじめ新たな情報・機能を追加しました。追加した機能により、世界中の研究者にとってより利便性が高く、また、情報も豊富となり、個別化医療・個別化予防などの次世代医療の実現に向け、着実な一歩となったと考えています。
主要なアップデート点の詳細
① INDEL頻度情報の追加
2018年6月に公開された3.5KJPNv2までは全て、一塩基置換によるSNV頻度が公開の対象となってきましたが、今回、塩基配列のINDEL(挿入(insertion, IN)・欠失(deletion, DEL))の頻度情報が追加されました。INDELは、例えばそれがタンパク質をコードする遺伝子上に存在した場合には、フレームシフトと呼ばれるアミノ酸配列への翻訳のされ方の大きなずれを生じ、時にそのタンパク質自体がつくられなくなるなど、大きな影響を及ぼすことが知られています。
○INDEL数(unfiltered)
| 総数 | うち新規数 | |
| 常染色体 | 7283994 | 4494320 |
| X染色体 (PAR1+PAR2) |
305476 | 197103 |
| X染色 (PAR1+XTR+PAR2) |
295683 | 186783 |
○INDEL数(フィルタをパスしたもののみ)
| 総数 | うち新規数 | |
| 常染色体 | 5839666 | 3295781 |
| X染色体 (PAR1+PAR2) |
244259 | 142720 |
| X染色 (PAR1+XTR+PAR2) |
240018 | 139221 |
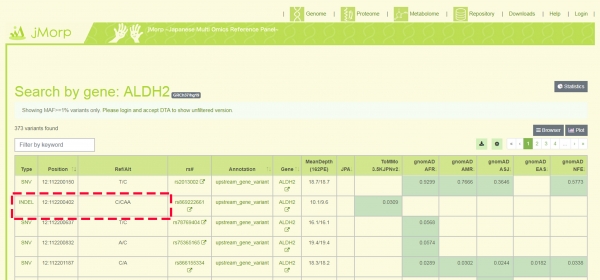
図1.頻度情報画面表示例(例:ALDH2)
② 1塩基ごとの平均深度データの追加
約500検体のマッピング結果から、1塩基ごとの平均深度及び深度の分散を算出しました。全ゲノム解析では、ゲノムの領域やその特性により、特にいわゆる短鎖型シークエンサーによって解読が難しいところ等があることが知られていますが、ToMMoによる解析によって、それぞれの領域がどのくらいの深度で解析されたかを示すデータを公開しました。これにより、各研究者が自身の興味・関心のある領域におけるToMMoのデータがどの程度の確度があるのかなど、詳細に知ることができます。
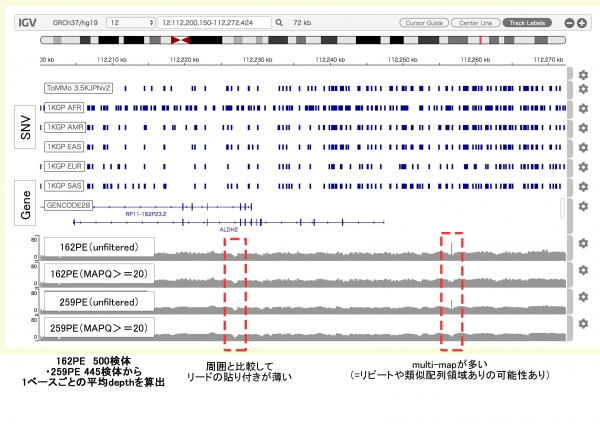
図2.Genome Accessibility dataset表示例
③ 変異情報をタンパク質立体構造上にマッピングするツールの追加
ゲノム上にある変異が、タンパク質をコードする遺伝子内にあり、且つそのタンパク質の立体構造がすでに解析されている場合に、その立体構造上での変異の位置をビューワーで見ることができます。問題となっている変異がタンパク質の構造の維持に影響しそうか、あるいは酵素活性などの機能に重要な影響がありそうか、などの推定の参考材料とすることができます。
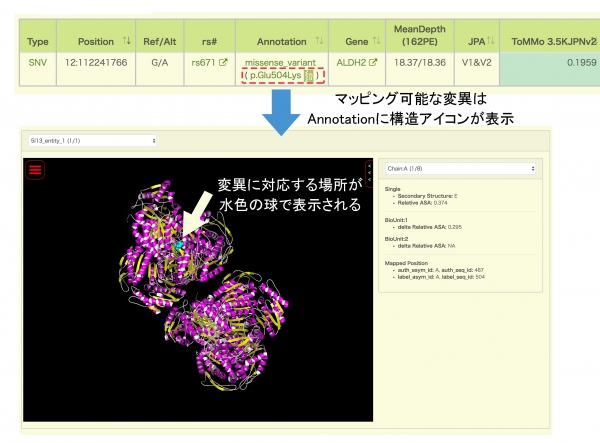
図3.変異の構造へのマッピング利用例
④ ゲノム解析レポジトリ情報のページの追加
今回、新たに4Kレポジトリ(WGS解析検体のサマリー)ページを追加しました。これまでToMMoでは、約3,500人の全ゲノム解析情報からSNVの頻度情報を公開し、また、解析結果を分譲対象としてきましたが、ToMMoで全ゲノム解析が完了している検体はそれ以上にあります。総計、4,007人についてのWGSに関する情報 (FASTQの塩基数、平均深度)及びその他の情報 (SNPアレイ解析情報、メタボローム解析情報) の有無について検索が可能となりました。
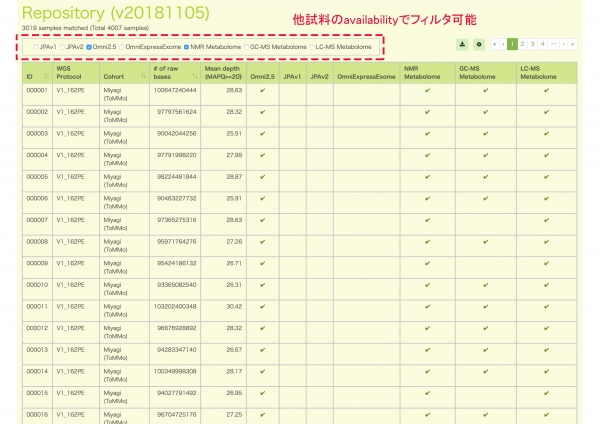
図4.4Kレポジトリ表示例
その他の新たな機能
gnomADの頻度情報データを表示する機能の追加
米国ブロード研究所が公開するデータベース「gnomAD」(約1.5万人の全ゲノム解析から検出した頻度情報が収録)の頻度情報を表示する機能が追加され、より多くの変異との頻度比較が可能となりました。
gnomAD URL:http://gnomad.broadinstitute.org
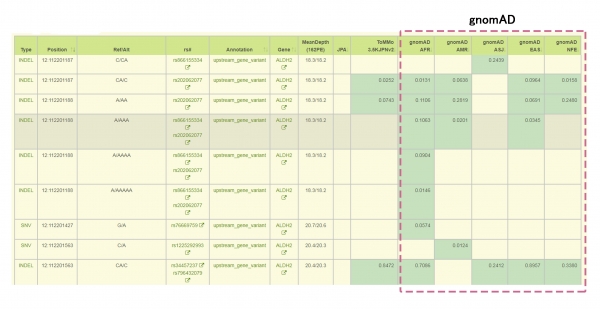
図5.gnomAD頻度情報表示例
