全ゲノムリファレンスパネルにMNV(multiple nucleotide variant)を追加
- 成果と活動
- これまでの成果の一覧
- 全ゲノムリファレンスパネルにMNV(multiple nucleotide variant)を追加
ToMMoはこれまで全ゲノムリファレンスパネルの人数規模を拡大するとともに、最初はSNV(single nucleotide variant、一塩基多型)のみだった搭載情報を、INDEL、HLA、CNV、STR、構造多型と次々に増やしてきました。
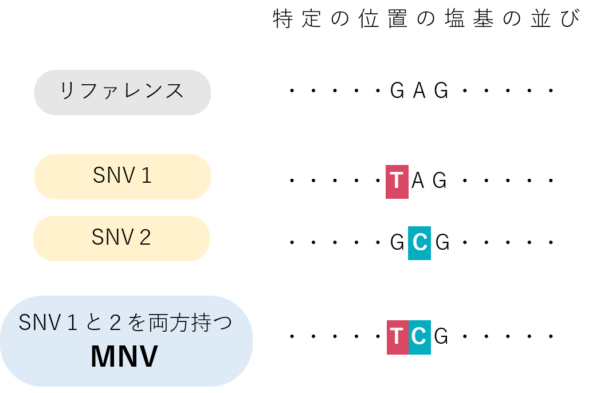
今回追加されたのはMNV(multiple nucleotide variant)の頻度情報です。MNVとは、近くに存在する複数のSNVの組み合わせのことで、この組み合わせが60KJPNでどういった頻度で存在するかを公開しています。
どうしてSNVだけではなく組み合わせであるMNVの頻度を公開したのでしょうか?
MNVの重要性を説明するために、私たちの経験を紹介しましょう。
特定のSNVを持っていると、大腸がんなどに罹りやすくなるリンチ症候群になることがわかっています。遺伝情報回付研究の一環でこのSNVを持つ人を54KJPNから抽出し、確認のために再度別の方法でゲノム解析を実施しました。すると、そのSNVの隣の塩基にも変異があった、つまりSNVが二つ並んでいたのです。よく調べてみると、実は隣のSNVはリンチ症候群を打ち消す働きがあることがわかりました。そして54KJPNから抽出した全員がこの二つ並んだSNV、つまりMNVを持っていたのです。(詳しく知りたい方はこちら)
SNVのみの解析では、色々な可能性を見落とすケースがある事が実感としてわかったため、今回新たにMNVの情報を加えることにしました。MNVパネルにより、変異の影響の理解が深まることが期待されます。
リンチ症候群の原因として検討したSNVは「ストップゲイン変異」でした。もっと長くアミノ酸が合成されるべきところ、この変異があると、この位置に終止コドンが生じて合成が止まってしまい、不完全なタンパク質になってしまうのです。ところが今回の場合、隣の塩基にも同時に変異があったため、終止コドンは生じずに別のアミノ酸をコードするようになる、つまり「ミスセンス変異」となっていました。その結果、別のアミノ酸への置き換えは生じるものの、アミノ酸の合成自体は途中で止まらないため疾患へのインパクトが下がったのです。今回の仕組みは、SNVの組み合わせであるMNVとして解析しないとわからないケースでした。
MNVは、今回の例のように疾患へのインパクトが下がる方向に働く場合もあれば、逆に上がる方向に働く場合もあります。
関連リンク
jMorpの全ゲノムリファレンスパネルおよびメタボロームデータを拡張
(2024年10月21日)