ゲノム構造多型解析結果「JSV1」を公開
- 成果と活動
- これまでの成果の一覧
- ゲノム構造多型解析結果「JSV1」を公開
ToMMoは、DNAの塩基配列を連続して長く読み取り可能な「長鎖リードシークエンサー」を用いた日本人のゲノム解析で、約 68,000ヶ所のゲノム構造多型を検出し、その頻度情報をデータベース「JSV1:Japanese Structural Variation」として公開しました。
ゲノムリファレンスパネルとゲノム解析情報を拡充~日本人全ゲノムリファレンスパネルが14KJPNに 構造多型データベースなど公開~【プレスリリース】
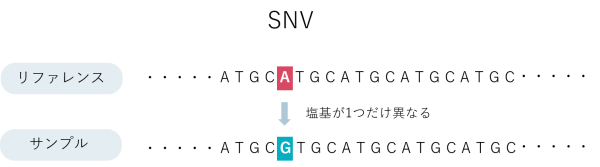
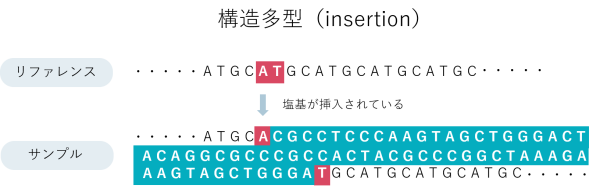
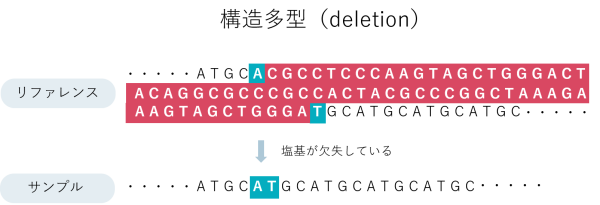
ゲノム多型とはゲノムの個人差で、他の人と異なっている配列のことを指します。これまでにToMMoが公開してきたゲノム多型は、SNV(配列が1塩基のみ異なる)と数十塩基の多型 (挿入<insertion>と欠失 <deletion>。まとめてINDELと呼ぶ) でした。
一方で、ゲノム多型の中には数百塩基〜数万塩基に及ぶものも数多く存在することがわかっており、こういった複雑で長い多型は「構造多型」と呼ばれています。
ToMMoでは、これまでゲノム解析の主流である短鎖リードシークエンサーで14,000人以上の日本人のゲノム配列を解析し、ゲノム多型の情報を全ゲノムリファレンスパネルとして公開してきました。しかし、短鎖リードシークエンサーでは、ゲノム構造多型が正確に検出できていないため、解析の対象はSNVや短いINDELに限定されていました。
短鎖リードシークエンサーによる解析の仕組みは(第3回 ToMMoではこうやってDNAを解読しています(前編:次世代シークエンサー))をご覧ください。
ただしゲノム構造多型もまた、個人の体質や疾患発症に影響していると考えられており、その解析は重要な課題です。
JSV1では、111トリオ (成人とその両親からなる三人組、合計333人) を解析し、構造多型を検出しました。そして、子にあたる成人が、両親の遺伝子を受け継いでいることを利用して精度検証を実施しています。ちょうど、親と子の解析結果を使って、お互いに答え合わせをするようなものですね。また、両親 (合計222人) の情報のみを用いて頻度情報を計算しています。これは、限られた数のデータで、一般集団のゲノム多型の頻度情報を計算するためには、血縁関係のある検体を除く必要があるためです。
長鎖リードシークエンス解析技術やゲノム構造多型の情報解析は、発展途上にあり、ToMMoではその解析技術の向上に取り組んできました。JSV1はその成果の第一歩です。今回の成果により、SNVや短いINDELの情報ではわからなかった疾患の原因が明らかになるかもしれません。

ゲノム解析に使用した長鎖リードシークエンサー
※多型とは集団で1%以上の個体が持っているバリアントを指すことが多いですが、本記事ではバリアント全体を多型としています。
(2022年1月21日)
